Status of this Document
All uppercase names like FIBER_UNIFORMARRAY_BASE_ATTRIBUTE are placeholders. They are expanded as stated in F5defs.h. In the final version of this document they will be replaced.
Introduction
F5 Coordinate Systems
F5 Chart Objects
F5 Topology Groups
- Provide unique dataspace information.
- Provide "indexDepth" information (vertex-related, cell-related, ...)
- Provide refinement levels
- (provide dimensionality information)
- Provide neighborhood information, either explicitely for unstructured grids, or implicitly from properties of any F5 Fields below.
F5 Representation Groups
F5 Fields
A field in F5 is the entity of all F5 Arrays, which are associated with the same field name. It may be represented by a single or multiple F5 Arrays. Multiple occurances are:
- in multiple timeslices
- in multiple grids
- in multiple topologies
- in multiple representations A field is uniquely specified by its name. All occurances of a F5 Array with the same name are part of the same field.
An optional reverse lookup information is provided via the /TableOfContents/ group and the /Fields/ group per Grid (RECOMMENDED).
F5 Arrays
- Note:
- To avoid ambiguous naming we use the name F5 Array instead of F5 Dataset. Otherwise it the difference between a HDF5 Dataset and a F5 Dataset might be obscured.
We're not very lucky with the notion Array.
More suggestions:
- F5 Values
- F5 Component
- F5 Brick
- F5 Block
- F5 Grain
- F5 Atom
Atom would be a good choice?
A F5 Array describes a multidimensional array of values. This might either be done by storing an explicit value at every node or by providing some kind of formular or building rule to generate the values. Providing building rules makes the structure of the array visible and could be useful for data compression. It might also be useful to optimize the file format for specific applications.
A F5 Array is represented in HDF5 by a HDF5 dataset with the name of the Field (contigous array), many HDF5 datasets within a HDF5 group named by the Field name together with special HDF5 attributes (fragmented array, direct product array or separated compound array) or a HDF5 group with special HDF5 attributes and no HDF5 datasets at all (uniform sampling, constant).
Information common to all F5 Datasets (except CONSTANT)
Each F5 Array provides
- Its type via an attribute FIBER_ARRAY_TYPE. The value of this type is an enum. The possible values are defined below at the descriptions of the various F5 Array types. It is represented as a string (hdf5 enum?). Right now, this attribute is only RECOMMENDED. It might change to REQUIRED in the future.
- Data Type information (also available for CONSTANT)
- If the F5 Dataset is an HDF5 dataset, then this is the HDF5 datatype
- If the F5 Dataset is represented by an HDF5 group, then the type is available via the type of the FIBER_HDF5_TYPEID_ATTRIB HDF5 or FIBER_UNIFORMARRAY_BASE_ATTRIBUTE HDF5 (MUST for linear map, otherwise MUST NOT) attribute. If FIBER_UNIFORMARRAY_BASE_ATTRIBUTE is not defined, then FIBER_HDF5_TYPEID_ATTRIB MUST be defined.
- Given an F5 Path object, the F5Fget_type() will provide an HDF5 type ID for a given F5 Dataset, regardless of its actual representation in HDF5 as group or dataset.
- The value of the FIBER_HDF5_TYPEID_ATTRIB is only used for constant datasets, otherwise its value may be arbitrary. RECOMMENDED is a null representation or a fill value.
SteffenProhaska proposes to REQUIRE a FIBER_HDF5_TYPEID_ATTRIB for simplicity. This would allow to check for the FIBER_ARRAY_TYPE and the FIBER_HDF5_TYPEID_ATTRIB in a generic way. This two information would carry a lot of human readable information without looking into the detailed structure of the specific F5 Array type.
- Data Space information: If the HDF5 object (group or dataset) contains an FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE attribute (n integers convertible to H5T_NATIVE_INT), then the dataspace is the multidimensional HDF5 simple dataspace constructed from this information. Otherwise, it is the dataspace of the first dataset found in the HDF5 group. Note that the FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE has precedence over an HDF5 dataset's dataspace. Example: An 1D dataset with an FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE of three integers is an three-dimensional F5 dataset.
- Note that the F5 Dataspace MUST be identical for all F5 Datasets within the same Topology group (except CONSTANT, see below).
Quick summary of this note: Werner und Steffen sind voellig unterschiedlicher
meinung. Werner propagiert, einen HDF5 Dataspace als die refrenz zu betrachten,
die die dimension des arrays bestimmt. Dadurch muss man integer indizes immer swappen.
Steffen ist der meinung, dass der HDF5 Dataspace eine nachgeordnete groesse ist und
das swappen der dimensionen nur einen notloesung ist, da hdf5 nicht genuegend features
bietet. Daher sollte immer explizit ein eigenes attribut gesetzt werden, dass
unsere absicht zum ausdruck bringt. Im moment scheint hier leider keine loesung
moeglich. Es koennte sein, dass die zukunft neue hdf5 features bringt, wobei unklar
ist, wie diese aussehen wuerden.
Ausfuehrliche diskussion:
Achtung, hier wird's kompliziert. Da koennen dann vielleicht auch mal die
anderen was dazu sagen, wie sie das alles verstehen:
Wenn man z.B. ein uniformes feld abspeichert, dann speichert man fuer die
positionen ein array mit float[3] oder besser struct { float x; float y; float z},
d.h. also z.B:
base = { 5, 7, 11 }, delta = {1, 2, 3} und die dimension in jede richtung, also
z.B. extent = { 3, 5, 7 }. Daraus berechnen sich die werte
pos = { (5, 7, 11), (6, 7, 11), ... (7, 15, 29) }
Und hier gehts schon los. Beim aufschreiben der pos wert habe ich Amira ordnung
angenommen. D.h. als erstes habe ich den x wert hochgezaehlt. Uebersetzt in
C/Fortran-array-order ist es FORTRAN order. Das kann man einsehen, wenn man
versucht die werte auf 'offensichtliche' weise in einem C array zu speichern,
das wuerde dann so aussehen:
position_t pos[dim_x][dim_y][dim_z];
In diesem falle wuerde aber im speicher nicht x zuerst gezaehlt, sondern z.
D.h. in amira ist das layout:
position_t pos[dim_z][dim_y][dim_x];
was FORTRAN oder entspricht. Man kann sich jetzt laenger darueber unterhalten,
was sinnvoller ist. Amira und viele andere, ich glaube auch Cactus, auch
ich sind der meinung dass fortran einfacher ist, da ich mit dem pointer auf
den ersten wert erst mal auf den ersten slice (x,y) zugreifen kann und die
dritte dimension erst dann dazu kommt, wenn ich sie brauche.
Jetzt kommt der punkt, der es kompliziert macht. hdf5 speichert datensaetze nur
in "C" order. D.h. ich kann den Dataspace eines amira datensatzes nicht einfach
auf einen hdf5 Dataspace abbilden. Dies geht nur, wenn ich die reihenfolge der
dimensionen umdrehe. D.h. der oben beschriebene uniforme koordinatensatz haette
den hdf5 dataspace {7, 5, 3}
Wir haben uns entschieden, dass F5/fiber erst mal alle datensaetze in FORTRAN
oder speichert, d.h. wenn man wirklich auf einen hdf5 dataset zugreifen will,
dann muss man jeweils die reihenfolge der indizes umdrehen.
Soviel zur einfuehrung in die problematik.
Ich bin jetzt der meinung, dass der F5/fiber dataspace erst mal nichts mit der
hdf5/C-Order/Fortran-Order zu tun haben sollte. D.h. ich finde, dass der F5
ArrayExtent in meinem beispiel {3, 5, 7} ist. D.h. ich nehme jeweils den ersten
wert aus all meinen vektoren mit rank 3 und mit jeweils diesem wert rechne ich
in der dimension, die x beschreibt, usw. Dass dann spaeter ein problem
auftritt, wenn ich dies als hdf5 datensatz speicher will, ist etwas anderes. Im
wesentlich handelt es sich hier um eine optimierung. Es ist einfach schneller
die indizes zu drehen, als jedesmal die daten von Fortran/Amira order auf
C-order umzusortieren. Jedoch erscheint es auch mir noetig an dieser stelle
explizit auf die problematik hinzudeuten.
Um verwirrungen zu vermeiden wuerde ich die ausdehnung des F5 arrays als extent
bezeichnen und dies als die entscheidende information betrachten. In meinem
obigen beispiel kommt ja gar kein hdf5 dataset vor und daher gibt es in diesem
fall eigentlich auch kein problem.
Wenn man jedoch daten auf diesem uniformen gitter speicher will, dann koennten
diese in einem hdf5 dataset gespeichert sein. Die groesse des F5 arrays waere
meiner meinung nach wieder {3, 5, 7} auch wenn der hdf5 dataset die groesse {7, 5, 3}
besitzt. In diesem fall koennten sehr schnell unklarheiten aufkommen.
Daher wuerde ich dafuer plaedieren auf jeden fall ein extent attribute an
jeden hdf5 dataset anzukleben und dann noch ein attribute, das auf die
storage order hinweist. Das koennte dann z.B. StorageOrder {2, 1, 0} sein.
Oder StorageOrder swapped, oder .... Da wir bisher vorschreiben, dass die
datensaetze in fortran order gespeichert sein muessen, darf es auch nichts
anderes sein. Es ist nur da, um auf die problematik hinzuweisen und um fuer
die zukunft gewappnet zu sein. Eine aktuelle F5 implementierung darf (MAY)
FORTRAN order annehmen, aber sollte (SHOULD) testen ob das auch stimmt.
Das was Werner oben vorschlaegt ist anders. Er schlaegt vor, dass der Dataspace
identisch mit dem hdf5 dataspace ist. D.h. auch wenn ich nur ein uniformes
gitter definiere, habe ich schon mit den gedrehten indizes zu tun, obwohl ich
glaube, dass es in diesem fall unnoetige komplexitaet ist.
Werner's Kommentar hierzu: Die Grundidee ist zu HDF5 selbst moeglichst wenig
hinzuzufuegen und dessen Funktionalitaet weitestgehend zu verwenden, dh. also
auch Attribute nur dort einzufuegen wo unbedingt noetig und die darin enthaltene
Information nicht auf anderem Wege ermittelt werden kann, zB. aus in HDF5 native
vorhandenen Strukturen. Im Falle der Dataspaces und Koordinaten zu jedem Punkt
ist die Vorgangsweise einfach: Genau dann wenn Indices auf physikalische
Koordinaten abgebildet werden muessen, dann muss gedreht werden, also index
0 ist z, index 1 ist y, index 2 ist x. Fallunterscheidungen wie oben sind
nicht noetig (wann drehen, wann nicht...). Solange man nur mit integer-
indices arbeitet, muss nie gedreht werden, erst bei einer Abbildung auf
{x,y,z}-phys. Koordinaten. Ob Drehen noetig ist oder nicht, ist somit
mehr eine Frage des Koordinatensystems (welcher Index hat welche
phys. Bedeutung) als des Datensatzes bzw. Dataspaces.
- Component Range information (RECOMMENDED) The FIBER_FIELD_COMPONENT_RANGE HDF5 attribute, two elements of the same type as the F5 dataset specifying the component-wise minimum and maximum of the data values in the dataset. Example: For the Positions field, these values denote the border of the bounding box. Implemented via F5Fget_range(), F5Fset_range().
- Component Average information (RECOMMENDED): The FIBER_FIELD_COMPONENT_AVERAGE HDF5 attribute, one element of the same type as the F5 dataset. Example: For the Positions field, this is the center of the bounding box. Implemented via F5Fget_average(), F5Fset_average().
- Component-wise Deviation information (RECOMMENDED): The FIBER_FIELD_COMPONENT_DEVIATION HDF5 attribute, one element of the same type as the F5 Dataset itself. The deviation provides new information. It could e.g. be the standard deviation of the data values.
Bevor nicht klar ist, welche groesse hier genau abgespeichert werden soll, wuerde ich es auf optional setzen. -- SteffenProhaska
- A descriptive comment telling the HDF5 representation type of the F5 dataset (contigous, constant, compound, fragmented, linear map, tensor product)
Contigous Array, FIBER_ARRAY_TYPE = "Contigous"
An HDF5 dataset with the same name as the F5 Array and the attributes
- FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE (OPTIONAL)
- FIBER_FIELD_COMPONENT_RANGE (RECOMMENDED)
- FIBER_FIELD_COMPONENT_AVERAGE (RECOMMENDED)
- FIBER_FIELD_COMPONENT_DEVIATION (RECOMMENDED)
Constant Datasets, FIBER_ARRAY_TYPE = "Constant"
An HDF5 data set of type H5S_SCALAR, specifying a single value. The Constant F5 dataset is special, in that it does not necessarily specify dataspace information by itself. Thus, it may be shared among multiple topologies (via HDF5 symlinks). In this case, it inherits the dataspace property from its respective parent topology (of the link, not of the actual location). The HDF5 dataset MAY possess an FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE attribute and is then restricted to topologies of the same F5 dataspace. A topology group MUST NOT be constructed only by constant datasets with no FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE attribute. F5 API: F5Lwrite_entity()
Separated Compound Type, FIBER_ARRAY_TYPE = "SeparatedCompound"
This F5 dataset type only makes sense for compound data types. There is no equivalent for scalar types. It is constructed by an HDF5 group with
- FIBER_HDF5_TYPEID_ATTRIB (MUST)
- FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE (MUST)
- For each component of the data type, a dataset named identically as the compound member, with the same type as the compound member. A certain dataset may be omitted, then this component of the compound is treated as beeing constant on the entire field. Its value is taken from the value of the FIBER_HDF5_TYPEID_ATTRIB attribute. If all components are present, then the value of the FIBER_HDF5_TYPEID_ATTRIB attribute is arbitrary.
Uniformly Sampled, FIBER_ARRAY_TYPE = "UniformlySampled"
An HDF5 group with the attributes
- FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE (MUST)
- FIBER_HDF5_TYPEID_ATTRIB (MAY)
- FIBER_UNIFORMARRAY_BASE_ATTRIBUTE (MUST)
- FIBER_UNIFORMARRAY_DELTA_ATTRIBUTE (MUST)
- no HDF5 datasets (MUST) The actual values $y$ are computed via a linear map of the kind
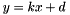 , where $x$ is a multidimensional index according to the parent topology's dataspace, k is the delta attribute and d is the base attribute.
, where $x$ is a multidimensional index according to the parent topology's dataspace, k is the delta attribute and d is the base attribute.
Direct Product Datasets, FIBER_ARRAY_TYPE = "DirectProduct"
These F5 Datasets only make sense for compound types. It is constructed by an HDF5 group with the attributes
- FIBER_FIELD_DATASPACE_DIMENSIONS_ATTRIBUTE (MUST)
- FIBER_HDF5_TYPEID_ATTRIB (MAY)
- FIBER_UNIFORMARRAY_BASE_ATTRIBUTE (MUST)
- FIBER_UNIFORMARRAY_DELTA_ATTRIBUTE (MUST)
- For each component of the data type, a dataset named identically as the compound member, with the same type as the compound member. There MUST be at least one such dataset.
- Note:
- F5 Datasets of this types are used for stacked coordinates.
- With no component datasets defined, they migrate to linear map datasets.
- If as many components are defined as compounds of the data member, then the values of the BASE and DELTA attribute are unused.
Fragmented Datasets, FIBER_ARRAY_TYPE = "Fragmented"
This F5 dataset denotes data values that are defined only on a subset of the complete F5 dataspace. They are represented in HDF5 by an HDF5 group named as the F5 dataset, with an arbitrary number of HDF5 subgroups or HDF5 subdatasets. The name of this sub-objects is arbitrary, but must be consistent among a topology group. Each such sub-object has the same structure as an F5 dataset by itself.
- each subobject (NOT the F5 dataset group itself) contains an FIBER_UNIFORMARRAY_BASE_ATTRIBUTE attribute of n-dimensional integer values specifying the integer origin of the first point of the datasets domain within the F5 dataset's dataspace.
- currently supported are
- contigous fragmented datasets
- linear map fragmented datasets
- separated compound fragmented datasets
- shall fragmented fragmented datasets be allowed via recursion?
 1.6.2
1.6.2